Générer le code LaTeX/chemfig d'une réaction chimique avec Zyme
Durant les trois années de licence bioinformatique, nous avons des cours de biochimie, et ceux ci viennent avec leur lots de structure chimiques à connaître.
En L1, j’avais réalisé un document pdf avec LaTeX/chemfig des acides aminées protéinogènes en représentation de FISCHER, et j’avais trouvé ça plutôt sympa, bien que ça m’avait pris pas mal de temps à rédiger.
Ajourd’hui, j’améliore ma méthode: fini le code de la structure en chemfig (extension LaTeX) a la mano, vive le code généré par du code !
Zyme
Zyme est un petit script python basé sur mol2chemfig, permettant à partir d’une représentation simplifiée d’une réaction chimique du type:
{Glucose} + ATP -> {Glucose-6-phosphate} + ADP + P_i
de générer le code chemfig correspondant, permettant le rendu de la structure chimique en utilisant la base de données PubChem.
Un fois que le fichier .tex est générer, il est possible de générer un pdf, ou un svg par exemple.
Installation
Rendez vous sur le dépôt git de zyme, et suivez les instructions d’installation.
Globalement, il suffit de cloner le dépôt et d’ajouter le dossier ./zyme au PATH de votre système (sous Linux). Je n’ai pas testé sous Windows, mais ça devrait fonctionner (🤞).
Utilisation
Pour générer le code LaTeX/chemfig d’une réaction chimique, il suffit de lancer la commande zyme avec en argument le fichier contenant la réaction chimique, et eventuellement le fichier de sortie.
Exemple:
zyme.py -i reaction.scheme -o reaction.tex
Le fichier reaction.scheme contient la réaction chimique au format texte, et le fichier reaction.tex contient le code LaTeX/chemfig généré.
Format du fichier de la réaction chimique
Le fichier de la réaction est un simple fichier texte contenant les réactifs, produits et flêches de la réaction chimique.
Un ‘;’ permet de séparer deux réactions différentes (plusieurs \schemestart[...]\schemestop seront générés).
Un mot entre accolades {} sera remplacé par le code chemfig de la structure correspondante (en faisant appel à l’API PubChem des formats SMILES).
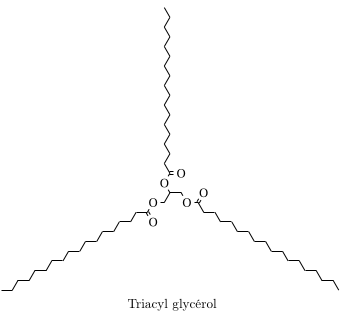
Exemple, pour le tryacilglycérol:
{triacylglycerol}
Donne dans un premier temps:
\schemestart
\chemfig{--[:60]--[:60]--[:60]--[:60]--[:60]--[:60]--[:60]--[:60]-(=[:300]O%
)-[:60]O--[:60](-[:120]O-[:60](-[:120]-[:60]-[:120]-[:60]-[:120]-[:60]%
-[:120]-[:60]-[:120]-[:60]-[:120]-[:60]-[:120]-[:60]-[:120]-[:60]-[:120])=O%
)--[:300]O-(=[:60]O)-[:300]--[:300]--[:300]--[:300]--[:300]--[:300]--[:300]%
--[:300]--[:300]}
\schemestop
Qui peut être compilé en pdf, en donnant:
Zyme accepte d’autres arguments, tel que +standalone pour générer un fichier .tex compilable directement (instruction \documentclass[]{standalone}).
Générer un .svg
Pour générer un fichier .svg à partir du fichier .pdf généré, il suffit d’utiliser la commande pdf2svg (à installer avec votre gestionnaire de paquet préféré).
pdf2svg reaction.pdf reaction.svg all
Conclusion
Et voilà, j’espère que ce petit outil vous sera utile.
N’hésitez pas à me remonter des bugs ou des suggestions d’améliorations, par exemple sur le dépôt git de zyme.